서 론
선택적 pre-mRNA 스플라이싱(splicing)은 단백의 다양성을 갖게 하는 전사 후 조절과정으로서, 스플라이싱 조절자 이상은 혈액 및 고형 종양 질환과 관련이 깊다[1].
Serine/arginine–rich (SR)과 family 단백의 일부인 SR splicing factor (SRSF) 1이나 SRSF3, SRSF6는 스플라이싱 조절자로 알려져 있고, 이들은 종양형성을 직, 간접적으로 관여하며, 이들 조절자의 돌연변이나 유전자 및 단백량의 이상은 다양한 종양성 질환에서 관찰된다[2]. SRSF1, SRSF2, SRSF3, SRSF6를 포함한 SR과 단백 및 heterogeneous nuclear ribonucleoprotein A1 (HNRNPA1)과 같은 스플라이싱 조절자들은 폐암, 난소암, 유방암 등의 다양한 암종에서 증가하며, 이들 중 SRSF1, SRSF3, SRSF6는 대장암에서 증가된다로 보고되었다[3,4]. 이외에 HNRNPA1과 같은 스플라이싱 조절자는 대장암의 잠재적인 진단적 표지자로서 보고되었다[4]. SRSF 단백들의 종양형성(oncogenic)에 관련된 중요한 역할과 임상적 유용성의 확립에 관한 많은 보고들에도 불구하고, 이들은 주로 SRSF1, SRSF3, SRSF6에 관련된 것이며, 아직 SRSF2와 SRSF4과 관련된 대장암 관련 종양형성 역할이나 발현 관련 연구는 상대적으로 드물거나 불분명하다.
SRSF2는 proto-oncogene의 동위형(isoform) 형성에 관여하며, SRSF2 단백 또는 유전자는 세포주기 진행세포증식에도 관여한다. 최근의 보고에 따르면 대장암에서 악성 예후에 관련되는 CD44의 v6 동위형의 발현을 촉진한다는 것이 있다[5,6]. 그외 자궁경부암을 유발하는 바이러스 유형인 유두종바이러스(human papillomavirus, HPV 16)의 oncoprotein 발현을 조절하며[7], 난소암에서도 SRSF2의 증가가 보고된 바 있다[8]. 또한 SRSF2 유전자 이상은 만성골수성단핵구성 같은 혈액종양에 발견되고 다양한 유전자의 단백발현을 조절한다[6]. SRSF1, SRSF2, SRSF3 및 SRSF6 등의 다양한 SRSF 단백들의 종양형성(oncogenic)에 관련된 중요한 역할과 임상적 유용성의 확립에 관한 많은 보고들에도 불구하고, SRSF4의 경우 골수구성백혈병에서 mRNA의 발현이 감소되고[9], 자궁경부암을 발생시키는 HPV type 16에서 발현이 증가한다는 보고가 있다[10]. 대장암과 연관하여서는 다른 splicing factor들에 비해 mRNA level이 감소하였다는 보고가 있으나[11] RSF4의 단백발현, 진단적 유용성 및 임상적 의미가 상세하게 밝혀지지는 않았다. 이들의 대장암과의 연관성을 확인하기 위해 Pubmed (www.ncbi.nlm.nih.gov)를 이용하여 ‘sc35 colon cancer’ ‘SRSF2 colon cancer’ ‘srp75 colon cancer’,’ SRSF4 colon cancer’ 등을 검색하였으나, protein level에서의 연구는 찾아보기 어려웠다.
스플라이싱 인자들을 임상에서 활용하기 위해서는 효과적인 표지자 선택과 표적 장기에서의 특성 규명이 필수적이다. 이전까지 이를 위한 연구들이 조금씩 진행되어왔으나, 대장암과 연관되어 진행된 것은 많지 않다. 최근 한국인 대장암 환자의 스플라이싱 인자의 발현을 알아보는 연구에서 SRSF3만이 암 조직에서 유효한 발현을 보였고, SRSF1, 5, 6, 7은 negative finding을 보였다[3]. 이에 본 연구에서는 남은 인자 중 SRSF2, 4에 대해 대장암에서의 단백 발현을 비교하고, 정상과 암 조직에서 이들 단백이 유의한 차이를 갖는다면 그 진단 및 임상적 우위를 비교하고자 하였다.
방 법
대상군 선정 및 검체 준비
원광대학교 병원 외과에서 2016년 8월부터 10월까지 대장암으로 수술을 받은 총 20명의 한국인 성인 환자들로부터 동의서를 취득한 후 얻어진 생검 샘플을 사용하였다. 각 환자의 분할조직(aliquot) 종양조직검체와 종양에 인접한 정상 양상을 보이는 분할(aliquot)점막 조직을 액체 질소에 보관 후 해동하여 분석하였다.
각 분할조직은 해동 후 포르말린으로 고정 후 파라핀을 침투시켜 포매 후 박절하고 헤마톡실린-에오신으로 염색하여 조직의 병리학적인 진단과 부합성을 확인하였다. 종양분할조직의 경우 50% 이상의 변성되지 않은 악성종양을 함유하고 있음을 확인하였다.
Immunoblot 및 반정량 분석
검체의 상태가 확인된 나머지 분할조직은 프로테아제(protease) 억제제를 함유한 RIPA buffer에 넣어 bullet blender 파쇄기(Next advance, Averill park, NY, USA)를 이용하여 균질화한 후 연속적인 원심분리를 이용하여 단백질을 얻었다. 검체당 50 μg의 단백을 10% sodium dodecyl sulfate-polyacrylamide 겔상에 분리하였다. 분리된 단백은 polyvinylidene difluoride 막으로 옮겨진 후 5% 탈지분유와 0.1% Tween 20 (TBS-T)함유한 Tris 완충식염수로 blocking한 후 1차 항체와 2시간 동안 반응되었다. 1차 항체는 다음과 같다: 항-SRSF2 (dilution 1:500; cat. 556363; Santa Cruz Biotechnology, Santa Cruz, CA, USA), 항-SRSF4 (Dilution 1:1,000; cat. HPA056975; Santa Cruz Biotechnology), 항-carcinoembryonic antigen (CEA) (Dilution 1:3,000; cat. MS-613-P, Thermo Fisher Scientific, Fremont, CA, USA), 항-HNRN-PA1 (Dilution 1:2000; cat. sc-32301; Santa Cruz Biotechnology).
단백을 포함한 막을 4회 세척 후, horseradish peroxidase가 결합된 2차 항체(anti-rabbit or anti-mouse)에 반응시킨 후 다시 4회 세척하였다. 막에 포함된 해당 단백 band는 Clarity Western ECL 기질액(Biorad, Hercules, CA, USA)을 이용하여 가시화하여 본 실험에서 정한 기준량에 대한 비율로 반정량 하였다. 각 검체의 광학적 밀도는 ImageJ (http://imagej.nih.gov/ij/)를 이용하여 분석되었다.
목표 분자량 영역에서 밴드가 검출되지 않는 경우는 SRSF2와 SRSF4는 0.8을, HNRNPA1과 CEA는 0.2로 간주하여 반정량하였다. 각 단백 밴드의 강도는 비정규 분포를 하였으므로 두 집단 간의 단백발현 정도의 비교는 비모수 통계 방법인 Mann-whitney U test를, 빈도 및 병기에 따른 차이는 chi-squared test로 MedCalc version 12.6.1.0 (Medcalc, Ostend, Belgium)을 사용하여 분석하였다.
결 과
환자군을 나이, 성별, TNM staging에 따라 분류하였다(Table 1). 평균 나이는 71.7세였고 60%가 남자였으며, 85%가 T3 이상, 35%가 N1 이상이었다. TNM staging에서 stage I으로 분류된 환자군이 15%, Stage II가 55%, stage III 이상은 30%를 차지했다.
Table 1.
Demographic characteristics of subjects and tumors
| Characteristic | Subject (n = 20) |
|---|---|
| Age (yr) | 71.7 (40–88) |
| Sex, male | 12 (60) |
| Tumor status | |
| T1/T2 | 3 (15) |
| T3/T4 | 17 (85) |
| LN metastasis | |
| N0 | 13 (65) |
| N1/N2/N3 | 7 (35) |
| TNM stage | |
| I | 3 (15) |
| II | 11 (55) |
| III/IV | 6 (30) |
스플라이싱 인자들과 CEA의 상대적인 단백정량 비교
SRSF2 (정상 4.0 vs. 종양 2.5, 종양은 정상의 0.625배, P=0.0449), SRSF4 (정상 1.5 vs. 종양 1.0, 종양은 정상의 0.667배, P=0.2428)는 정상 조직에서 종양 조직에 비해 단백 발현이 낮은 경향을 보였으며, 특히 SRSF2는 통계학적 의의를 보였다. 반면, HNRNPA1 (정상 0.8 vs. 종양 3.0, 종양은 정상의 3.75배, P=0.0005)과 CEA (정상 0.8 vs. 종양 2.0, 종양은 정상의 2.5배, P<0.0001)는 종양 조직이 정상 조직보다 현저하게 발현이 증가되어 있었다(Fig. 1).
Fig. 1.
Protein levels of splicing factors and carcinoembryonic antigen (CEA) in Korean patients with colon cancer. (A) Semiquantitative immunoblot analysis of colon cancer (CA) and adjacent normal-appearing (NR) samples. The β-actin (ACTB) band was used to confirm the protein loading quantity. The study-defined standard cell lysate (<120 µg of protein mixture derived from CA and NR samples) was loaded as a semiquantitative calibrator (S1, S2, and S3). For each serine/arginine-rich splicing factors (SRSF) and CEA, distinct bands in the following target molecular weights were analyzed: 30–40 kDa (SRSF2), 44–66 kDa (SRSF4), 25–35 kDa (HNRNPA1), and 180–220 kDa (CEA). (B) Relative levels of splicing factors and CEA. Each bar indicates the median and the black horizontal lines indicate the interquartile ranges. HNRNPA1, heterogeneous nuclear ribonucleoprotein A1.
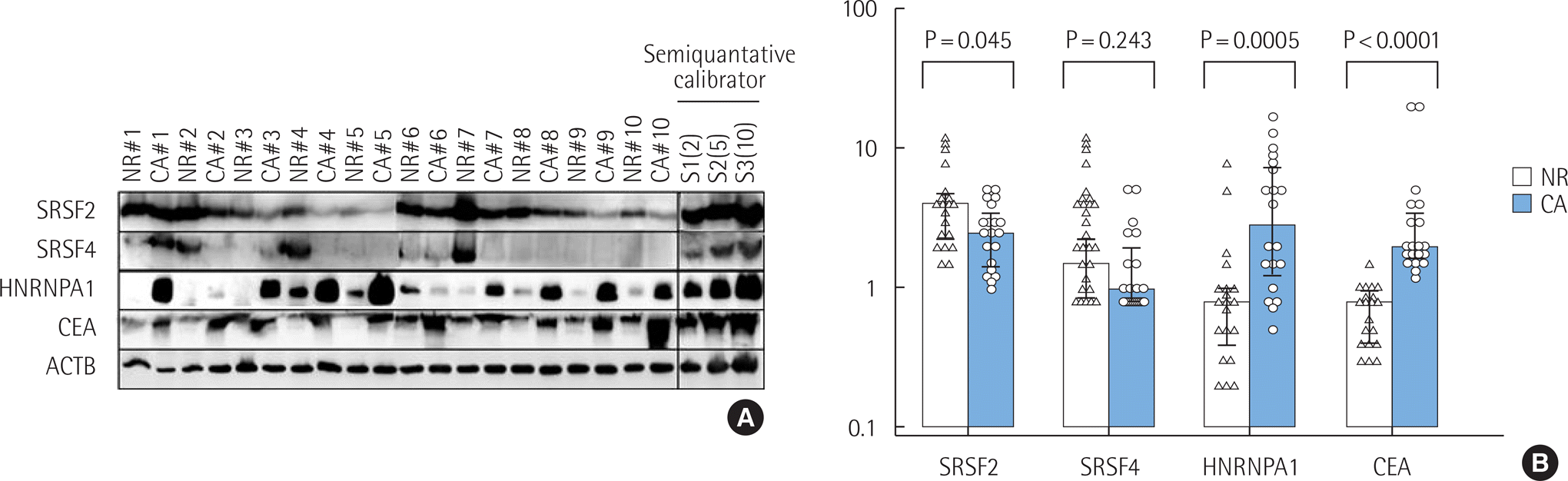
동일한 환자에서 얻어진 종양과 정상 조직을 쌍으로 비교한 결과(Table 2) 종양에서 정상 조직에 비하여 SRSF2가 2배 이상 발현이 증가한 빈도는 없었으며, 25% (5/20)에서 정상 조직 발현이 더 높은 빈도를 보였다. SRSF4가 종양에서 정상 조직에 비하여 발현이 증가한 빈도는 10% (2/20)였고, 35% (7/20)에서는 정상조직에서 발현이 높았다. 반면, HNRNPA1 (65%)와 CEA (80%)는 대부분의 경우에서 종양 조직에서 정상 조직보다 2배 이상 단백발현이 높았으며 모든 경우에서 P-value는 0.05 이하로 통계학적 의의를 보인다.
Table 2.
Comparison of upregulation incidences of splicing factors and CEA in Korean patients with colon cancer
| Upregulated (≥ 2-folds) incidence in paired samplesa) | Increased in cancer tissue | Increased in normal tissue | P-valueb) |
|---|---|---|---|
| SRSF2 | 0/20 (0) | 5/20 (25) | 0.027 |
| SRSF4 | 2/20 (10) | 7/20 (35) | 0.02 |
| HNRNPA1 | 13/20 (65) | 2/20 (10) | 0.0001 |
| CEA | 16/20 (80) | 0/20 (0) | < 0.0001 |
병기에 따른 스플라이싱 인자의 차이를 알아보기 위해 TNM stage I/II인 14 case와 Stage III/IV에 해당하는 6 case를 비교한 결과, SRSF2와 SRSF4뿐 아니라 HNRNPA1, CEA 모두 통계학적으로 의미 있는 결과가 도출되지 않았다(P-value 각각 0.7237, 0.41, 0.076, 0.1573).
고 찰
최근 많은 보고들에서 스플라이싱 인자의 종양형성의 중요성과 진단적 표지자로서의 유용성 규명에도 불구하고, SRSF2와 SRSF4의 대장암에서 단백발현 특성에 대한 보고는 찾아 보기 어렵다.
본 연구에서는 한국인의 대장암 환자의 암 조직에서 이들 두 단백의 발현은 정상 조직에 비해 감소되는 모습을 보았으며, 동시에 대장암 검출에서 기존 대장암 표지자인 CEA의 우월성을 확인하였다. SRSF2의 종양에서의 중요한 역할 규명과 함께 SRSF2 단백의 증가는 91%의 상피세포성 폐암 및 89%의 신경내분비계 폐암에서 보고된 바 있으나[12,13], 위장관 종양에서 SRSF2의 발현이 감소된 보고도 있다[14]. 본 연구에서는 대장암 조직에서의 SRSF2 발현의 중앙값과 증가한 빈도에서 대장암 조직에서의 SRSF2 발현이 통계적으로 의의 있게 감소됨을 보인다.
본 연구의 결과는 급성 골수구성 백혈병에서 SRSF2 유전자 발현의 증가보다는 감소를 보인 보고들과 일치하는 경향을 보이며[9], 또한 폐 선암 및 편평세포암에서 보인 현저한 SRSF2 단백증가 결과와는 배치되는 모습을 보인다[12]. 상기 연구들과의 차이는 장기별 조직학적 특성, 혹은 연구에 사용된 항체의 항원 인식부 결합능에 의한 방법적 차이 등을 고려할 수 있으며, 이는 많은 종양표지자에서 흔히 관찰되는 모습이다.
siRNA에 의한 SRSF4 유전자의 감소는 cisplatin과 같은 항암제에 의한 세포죽음을 막는다고 보고된 바 있으며[15], 이는 SRSF4가 항암제에 의한 세포죽음을 촉진하는 방향으로 유도하고, 유도할 가능성이 높음을 제시한다. 이러한 관점에서 암세포에서 SRSF4는 정상보다 감소할 가능성이 높으며, SRSF4가 종양조직에서 발현이 증가된 대부분의 SR 스플라이싱 인자들과 반대의 작용을 할 수 있는 가설을 제시할 수 있다. 본 연구 역시 언급한 가설과 부합하는 소견을 보인다. 이외에 급성골수구성백혈병에서 SRSF4 유전자의 감소를 관찰한 결과나[9] 신장암세포에서 이 유전자의 증가반응을 관찰하지 못한 소견[16]과 부분적으로 일치한다.
HNRNPA1과 CEA는 기존에 보고된 것처럼 본 연구에서도 대장암에서 그 발현이 현저히 증가되었고, 그 빈도가 매우 높아 대장암의 진단적 표지자로서 유용성을 재확인 할 수 있었다. 특히 HNRN-PA1는 암세포의 대사와 관련되며, 세포고사를 막는 인자로도 알려진 바 있다. 일부 간암 연구에서 스플라이싱 인자는 환자의 예후나 병기에 관계된다는 보고가 있다[17].
본 연구에서 Stage III 이상의 높은 병기의 환자들이 낮은 병기의 환자들보다 현저하게 높은 스플라이싱 인자의 발현을 보이지는 않아 병기와 상관성은 없는 것으로 추정되었다. 그러나 본 연구의 증례가 적어 추가적인 연구가 필요할 것으로 판단된다.
한국인 대장암 환자의 암 조직에서 SRSF2와 SRSF4는 정상 조직에 비해 대장암 조직에서의 검출빈도는 낮았으며, 제한된 수이기는 하나 높은 병기와도 주목할 만한 양의 상관성을 보이지 않았고, 기존 표지자인 CEA 등에 비해 뚜렷한 우월성을 보이지도 않았다. 때문에 임상에서 즉각적인 종양 표지자로서의 이용에는 더 많은 연구가 필요할 것으로 생각되며, 아직은 기존 표지자를 사용하는 것이 추천된다.





